四大皮肤生理区域上的微生物
首先,我们来看下这张图。科学家根据人体生理特征,将皮肤主要划分成了四个区域。①分别是,油性区域、湿润区、干性区域以及足部区域。
②位于皮肤上的微生物,整体来看,主要有细菌、真菌和病毒。
③真菌在四个区域的分布多样性较为简单,主要以马拉色菌为主。
④相对于真菌,细菌的多样性更加丰富,在各部分不同细菌占比也有所不同。主要以丙酸杆菌、棒状杆菌和葡萄球菌为主。
⑤从个体上看,不同的人携带的微生物也有所差异。主要由于先天原因基因、种族等导致,以及后天环境等因素共同作用导致的。
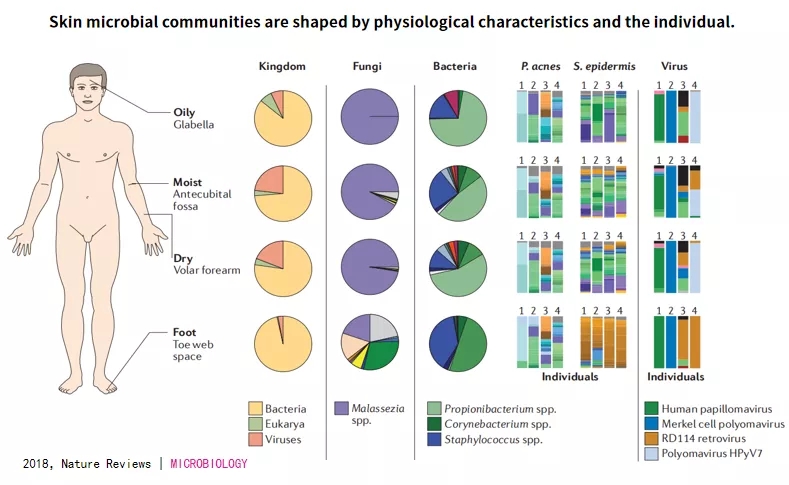
从这张图,我们看出,皮肤微生物的组成主要由皮肤不同部位的生理特征和个体差异共同决定。
四大区域皮肤微生物前十名汇总表
这张表总结了位于四大皮肤区域,排名前十的微生物菌种。上面是细菌的总结,下面是真菌的总结。油性部位主要以丙酸杆菌为主,而湿润部位,主要以葡萄球菌和棒状杆菌为主。而真菌则是以马拉色菌为主。

这里就要说到,达尔文的“物竞天择,适者生存”进化论了,微生物在长期的磨练下,进化出了各种强大的生存能力与价值。
微生物的进化和语言密码
这些微生物不仅能分泌附着物,促进自身粘附皮肤;并且利用皮肤上的代谢物,为皮肤进行清洁的同时,还能为皮肤提供营养和保护。曼曼君带你来具体看下他们的强大生存技能。
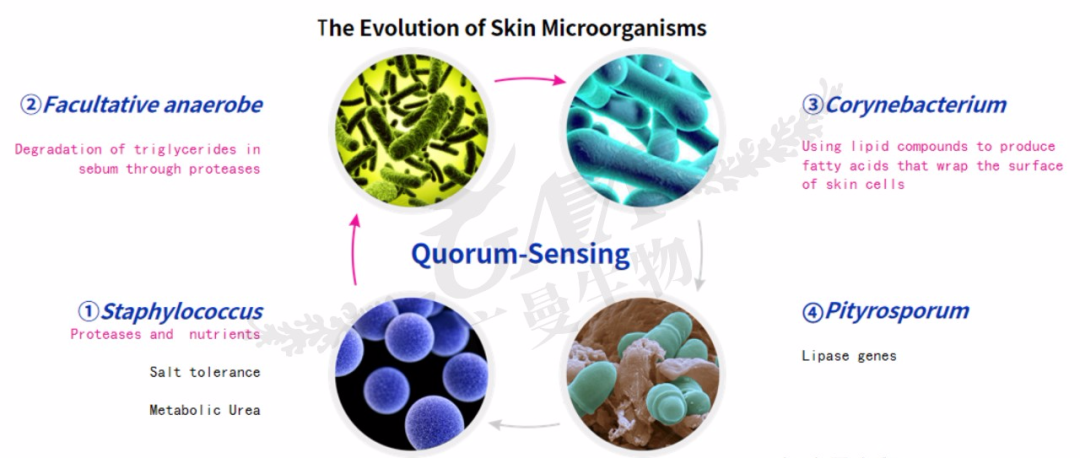
从整体看,微生物之间通过细菌群体感应进行宏观调控。细菌群体感应可从以下几方面进行理解:①细菌产生并释放特定信号分子,感知其浓度变化并协调群体行为,这一调控系统被称为群体感应(QS);②信号分子达到临界浓度后,细菌启动特定基因表达,群体开始发挥整体作用;③QS是动植物共生菌群的常见现象,深入研究QS的机制对致病菌等相关研究非常重要;④通过理解QS的遗传、基因组、生物化学和信号分子的多样性,人类开始充分认识QS和细菌社会性之间的联系等。
细菌群体感应,比人类语言更为复杂,不仅能够调控群体间的互动,并且,还能进行菌群生长的自我调节,类似于我们人类社会的计划生育。当细菌数目超出皮肤的承载能力时,就会释放一些信号因子,下游基因接收到这些信号,就会暂时关闭对应基因的表达和蛋白的合成,从而控制菌群的过度繁殖。目前科学界对细菌群体感应机制尚不完全清楚,仍处于在探索之中。

曼曼君从微生物的进化中也得到了许多感悟:蜕变从来就不是轻松的事情,但是可以做到努力不辜负自己。世界也本没有完美,但努力可以接近完美!比如,微生物经过上亿年的进化都逐渐找到了最适合自己的生存模式。屏幕前的你,如果遇到了生活或者工作中上的挫折时,也可以告诫自己再努力一次:微生物都这么努力,你有什么理由做咸鱼?——那就再努力一次!
皮肤微生物在缺乏营养的皮肤表层更加成就了它强大的生存功能。这也说明好的木材并不在顺境中生长,风越强,树越壮。

我们立志打造守诚信、重品质、敢担当、谋共赢的商业文明。我们不断创新,致力于开发出更多满足市场需求的新型防腐方案。广曼成立至今,一直致力于为日化行业提供更安全、更高效、更可持续的个人护理防腐方案。
随着越来越多的化妆品法规发布,对日化行业的要求也越来越高。广曼愿与您携手共进,打造质量过硬的优质产品,共创中国民营企业商业文明!
参考文献:
1. Bai X, She Y. Selective epoxidation of linear terminal olefins with metalloporphyrins under mild conditions[J]. Frontiers of Chemical Engineering in China, 2009, 3(3): 310-313.
2. Chen X, Ning X, Kang Y, et al. Aerobic Acetoxyhydroxylation of Alkenes Co-catalyzed by Organic Nitrite and Palladium[J]. Organic Letters, 2016, 18(20): 5368-5371.
3. Jia W, Li H, Zhao L, et al. Gut microbiota: a potential new territory for drug targeting[J]. Nature Reviews Drug Discovery, 2008, 7(2): 123-129.
4. Michael B, Doron S, Shiri M. Microbial biofilms and the human skin microbiome[J].npj Biofilms and Microbiomes 2016; 2(1):10.1038/s41522-016-0004.
5. Byrd A L, Belkaid Y, Segre J A, et al. The human skin microbiome[J]. Nature Reviews Microbiology, 2018, 16(3): 143-155.18)
6. Lita M P, Heather H C, Jennifer M F, et al.The Integrative Human Microbiome Project. Nature 2019: 10.1038/s41586-019-1238-8






 2021-06-13
2021-06-13 加载中...
加载中...









